H τεχνητή νοημοσύνη της Google που έγινε πρωτοσέλιδο όταν απέδειξε ότι μπορεί να προβλέπει τις δομές πρωτεϊνών τίθεται στη διάθεση του μεγαλύτερου εργαστηρίου βιολογίας στην Ευρώπη, με στόχο να δημιουργηθεί βάση δεδομένων που θα καλύψει το σύνολο των ανθρώπινων πρωτεϊνών.
Η DeepMind, λονδρέζικη εταιρεία τεχνητής νοημοσύνης που εξαγοράστηκε από τη Google, συνεργάζεται στο άκρως φιλόδοξο πρόγραμμα με το Ευρωπαϊκό Εργαστήριο Μοριακής Βιολογίας (EMBL), με έδρα τη Χαϊδελβέργη της Γερμανίας.
Η συνεργασία «αντιστοιχεί στην σημαντικότερη συνεισφορά της τεχνητής νοημοσύνης στην πρόοδο της επιστημονικής γνώσης» σχολίασε ο κυπριακής καταγωγής Ντέμις Χασάμπις, ιδρυτής και διευθύνων σύμβουλος της DeepMind.
Η εταιρεία του έχει ήδη δημιουργήσει τη μεγαλύτερη βάση δεδομένων με δομές ανθρώπινων πρωτεϊνών, η οποία περιλαμβάνει μέχρι στιγμής 350.000 καταχωρήσεις.
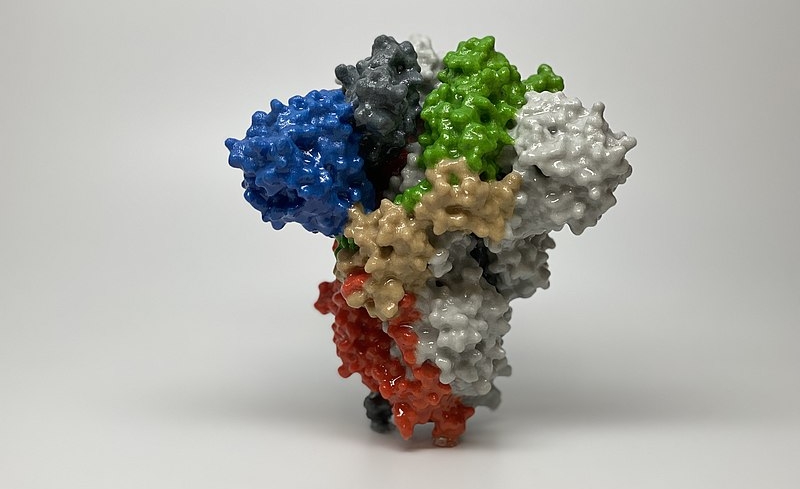
Μοντέλο της πρωτεΐνης-ακίδας του κορωνοϊού. Το σχήμα της τής επιτρέπει να συνδέεται στα ανθρώπινα κύτταρα όπως το κλειδί στην κλειδαριά (US NIAID)
Τίποτα δεν λειτουργεί σωστά στα κύτταρα χωρίς τις πρωτεΐνες, μακριές αλυσίδες από αμινοξέα που αναδιπλώνονται σε περίπλοκα, τρισδιάστατα σχήματα. Το σχήμα κάθε πρωτεΐνης είναι αυτό που καθορίζει τον τρόπο που δρα, είτε πρόκειται για δομικές πρωτεΐνες είτε για ενζυματικές πρωτεΐνες που ρυθμίζουν τον μεταβολισμό.
Γι΄αυτό και η δυνατότητα πρόβλεψης της δομής τους αναμένεται να παίξει σημαντικό ρόλο στη βιολογία, τη βιοχημεία και τελικά την ιατρική.
Μέχρι σήμερα, η τρισδιάστατη δομή μπορούσε να υπολογιστεί μόνο με απαιτητικές και άκρως χρονοβόρες μεθόδους όπως η κρυσταλλογραφία ακτίνων Χ και η κρυοηλεκτρονική μικροσκοπία.
Η κατάσταση άλλαξε με θεαματικό τρόπο τον Νοέμβριο, όταν η λονδρέζικη εταιρεία DeepMind παρουσίασε το AlphaFold, μια τεχνητή νοημοσύνη που προβλέπει με σχετική ακρίβεια την τρισδιάστατη δομή πρωτεϊνών γνωρίζοντας μόνο την αλληλουχία των αμινοξέων της (η οποία καθορίζεται από το αντίστοιχο γονίδιο).
Το AlphaGo νίκησε κατά κράτος στον διεθνή διαγωνισμό αναδίπλωσης πρωτεϊνών CASP, ο οποίος διεξαγόταν για χρόνια χωρίς μεγάλα βήματα προόδου.
Η ανακοίνωση του ΕΜΒL για τη συνεργασία με την DeepMind έρχεται λίγες ημέρες μετά την παρουσίαση της τεχνολογίας του AlphaGo στο περιοδικό Nature.
Το AlphaFold είναι πλέον διαθέσιμο ως λογισμικό ανοιχτού κώδικα, κάτι που θα επιτρέψει σε ερευνητές σε όλο τον κόσμο να πειραματιστούν μαζί του και να το βελτιώσουν.
Ελεύθερη θα είναι η πρόσβαση και στη νέα βάση δεδομένων που αναπτύσσει το EMBL, η οποία θα περιλαμβάνει σε πρώτη φάση τις τρισδιάστατες δομές 350.000 πρωτεΐνών.
Η βάση δεδομένων (AlphaFold Protein Structure Database) σταδιακά θα επεκταθεί στα 130 εκατομμύρια τρισδιάστατα μοντέλα, αναφέρει το EMBL στην ανακοίνωσή του. O αριθμός αυτός είναι 700 φορές μεγαλύτερος σε σχέση με τις καταχωρήσεις του Protein Data Bank, της μεγαλύτερης αντίστοιχης βάσης μέχρι σήμερα.
Και, εκτός από τις ανθρώπινες πρωτεΐνες, το μεγάλο ερευνητικό πρόγραμμα θα καλύψει και πρωτεΐνες 20 ακόμα οργανισμών μεγάλης βιολογικής σημασίας, όπως το παράσιτο της ελονοσίας και το βακτήριο της φυματίωσης.